目录
- UTGDiff 是一款能直接读取化学家文字指令,并将其翻译成新分子结构的 AI,让按需设计分子成为可能。
- AlphaFold3 迎来一次进化,首次将非蛋白分子纳入预测。但 CASP16 的官方结果表明,它在复合物、RNA 和硬靶点上并非无敌,老方法配合聪明策略依然能打。
- ESMDynamic 学习了海量蛋白质「电影」,仅凭单条氨基酸序列,就能快速预测蛋白质的动态行为。这项任务过去需要超级计算机才能完成。
1. AI 按指令造分子:从文本到图谱
让计算机设计分子,常常面临沟通障碍。化学家想下达这样的指令:「设计一个分子,包含一个吡啶环,通过酰胺键连接到一个噻唑环上,且溶解度要好。」但 AI 无法直接理解。过去的工具要求使用 SMILES 这类机器语言或复杂的参数来约束生成过程,如同要求建筑师依据砖头数量而非设计蓝图来盖房子。
过去的 AI 模型只能匹配关键词,无法理解深层设计意图。就像你告诉它「我想要个蓝色的、四个轮子的东西」,它可能会给你一辆蓝色的购物车。
北京大学和香港大学的一项新研究,让这个只会匹配关键词的 AI 学会了读懂设计蓝图。
AI 是怎么学会「看图说话」的?
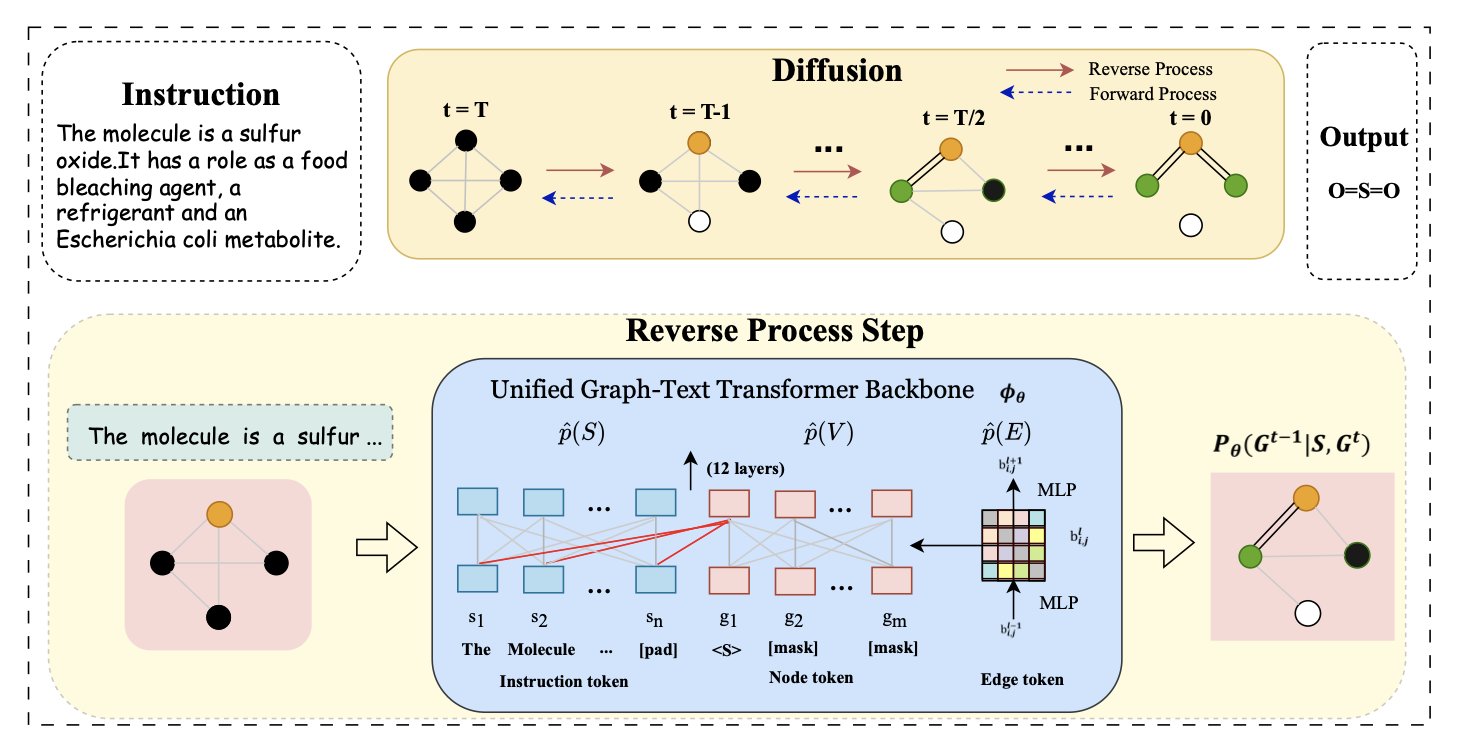
新模型名为 UTGDiff。它没有将理解文本和生成分子图谱作为两个独立任务再强行拼接。它将二者置于一个统一的框架内,使其在整个生成过程中持续「对话」。
UTGDiff 采用一种称为扩散模型 (Diffusion Model) 的方法。这个过程好比修复一张充满噪点的旧照片。初始状态是一张模糊的分子图谱,由随机的原子和化学键构成,同时还有一条清晰的文字指令,例如「我想要一个带吡啶环的噻唑」。
AI 的任务就是逐步去除这张图谱上的噪点。关键在于,在去除噪点的每一步,AI 都会参照那条文字指令,判断当前操作是否让分子更接近指令描述的结构。
这种在生成过程中持续、细粒度的文本指导,是 UTGDiff 的核心机制。它在生成的每一步都用文本指令校准方向,确保过程的准确性。
结果怎么样?
当 AI 学会参照指令来构建分子后,生成结果的质量大幅提升。
在多项任务中,UTGDiff 的表现超越了现有方法。它生成的分子与人类指令的相似度更高,化学有效性也好,产出的不是无效的随机组合。
UTGDiff 还具备出色的「零样本」 (Zero-shot) 能力。面对训练中未见过的新指令,它也能理解并执行,无需额外训练。
这项工作改变了分子生成的方式,我们可以开始尝试将脑中的构想直接告知机器,并观察其转化为真实的分子结构。
📜Title: Instruction-Based Molecular Graph Generation with Unified Text-Graph Diffusion Model
📜Paper: https://arxiv.org/abs/2408.09896
💻Code: https://github.com/ran12/UTGDiff
2. AlphaFold3 在 CASP16:进化了,但还没成神
AlphaFold3 (AF3) 发布后,赞誉与质疑并存。现在,权威的 CASP16 结构预测竞赛公布了结果,让我们可以在同一个赛场上,检验 AF3 的真实水平。
最大的变化:从专科医生到全科医生
AF3 相较于 AF2 最大的进步,是它不再局限于蛋白质。它学会了处理小分子、核酸(RNA)、离子等。对药物发现领域,这简化了流程。过去用 AF2 预测靶点结构,还需用另一套软件预测药物分子如何结合。AF3 将这两步合二为一。
可以这样理解:AF2 像个顶级的蛋白质木匠,手艺精湛,但只做木工。AF3 则像个总承包商,除了木工,还能处理水电和装修。整个流程因此简化,对非计算专家也更友好。
硬碰硬:它真的比 AF2 强很多吗?
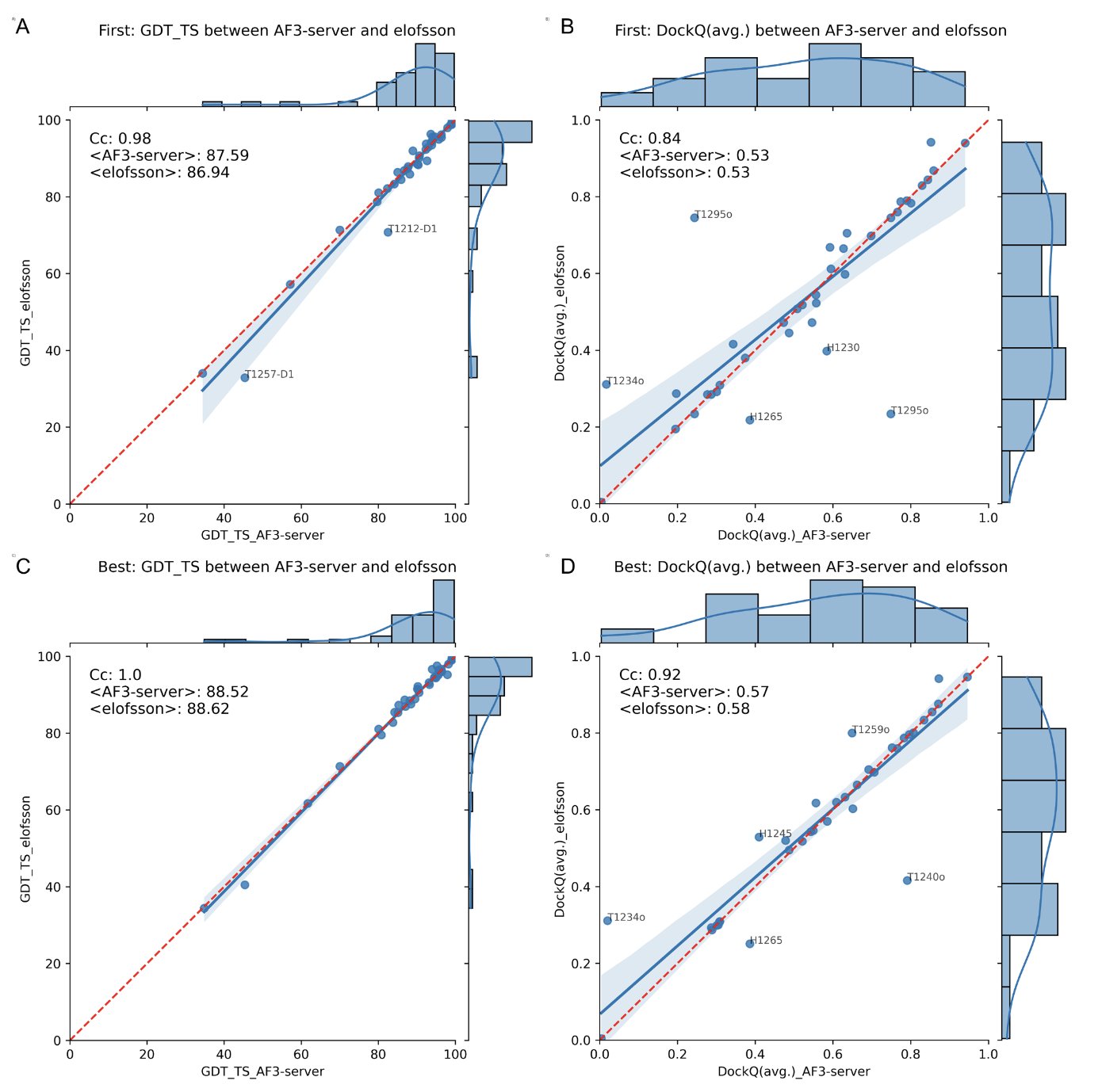
AF3 比 AF2 强,但优势有限。
在预测蛋白质复合物时,AF3 的表现略好于 AF2。但一个有趣的发现是,如果给 AF2 足够的计算资源进行「海量采样」(即穷举试错),其最终表现与 AF3 并无差异。
这表明 AF3 在蛋白质结构预测的核心引擎上,可能没有突破性突破。它的优势更多体现在整合与易用性,而非绝对的预测精度。
裂缝出现:AF3 在哪里会翻车?
任何工具都有边界,AF3 也不例外。CASP16 的结果指出了它的几个弱点。
首先是复合物的组成(stoichiometry)。AF3 在预测多亚基蛋白复合物时,常会搞错各个亚基的数量。它能画出漂亮的引擎,却可能漏掉几个活塞。对于理解生物机器的运作,这是个根本性缺陷。
其次是RNA 结构。RNA 像煮过的面条,柔软无定形,其结构预测向来是难题。AF3 作了尝试,结果尚可,但远未达到其在蛋白质预测上的统治地位,甚至未能在此单项拔得头筹。
最后是高难度靶点。对于结构新颖、缺少同源蛋白参考的「硬骨头」,AF3 的表现并未超越其他顶尖方法。在探索生物学的未知领域时,仍需依赖多种工具与人类智慧,而非单一的「万能神器」。
新的挑战:怎么从一堆「正确答案」里挑出最好的?
和 AF2 一样,AF3 也不会只生成一个答案,而是输出一堆看似合理的模型。新问题随之而来:如何从中选出最接近真实结构的模型?CASP16 的报告也强调,结构预测能力在飞速提升,但评估和筛选最佳模型的能力却相对滞后。
AF3 是一项了不起的工程成就。它更通用、更易用,进一步降低了结构预测的门槛。但它并非魔法,也不是结构生物学的终点。难题依然存在,旧问题解决了,新问题又浮现。
📜Title: AlphaFold3 at CASP16
📜Paper: https://onlineliibrary.wiley.com/doi/epdf/10.1002/prot.70044
3. ESMDynamic:单序列预测蛋白动态
AlphaFold 如同一个高精度「照相机」,能拍出蛋白质清晰的静态「证件照」。但蛋白质在细胞内是动态的,像一台持续扭动、折叠的复杂机器。药物分子常常结合于静态「证件照」中无法捕捉的瞬时「激活」构象上。
要观察蛋白质的动态,需要借助分子动力学(MD)模拟。MD 像一台拍摄分子世界「电影」的摄像机,但它耗能巨大且拍摄缓慢。获得一段有意义的动态影像,往往需要一个计算集群运行数周。
ESMDynamic 提供了一个「快进」工具。
AI 的学习方式
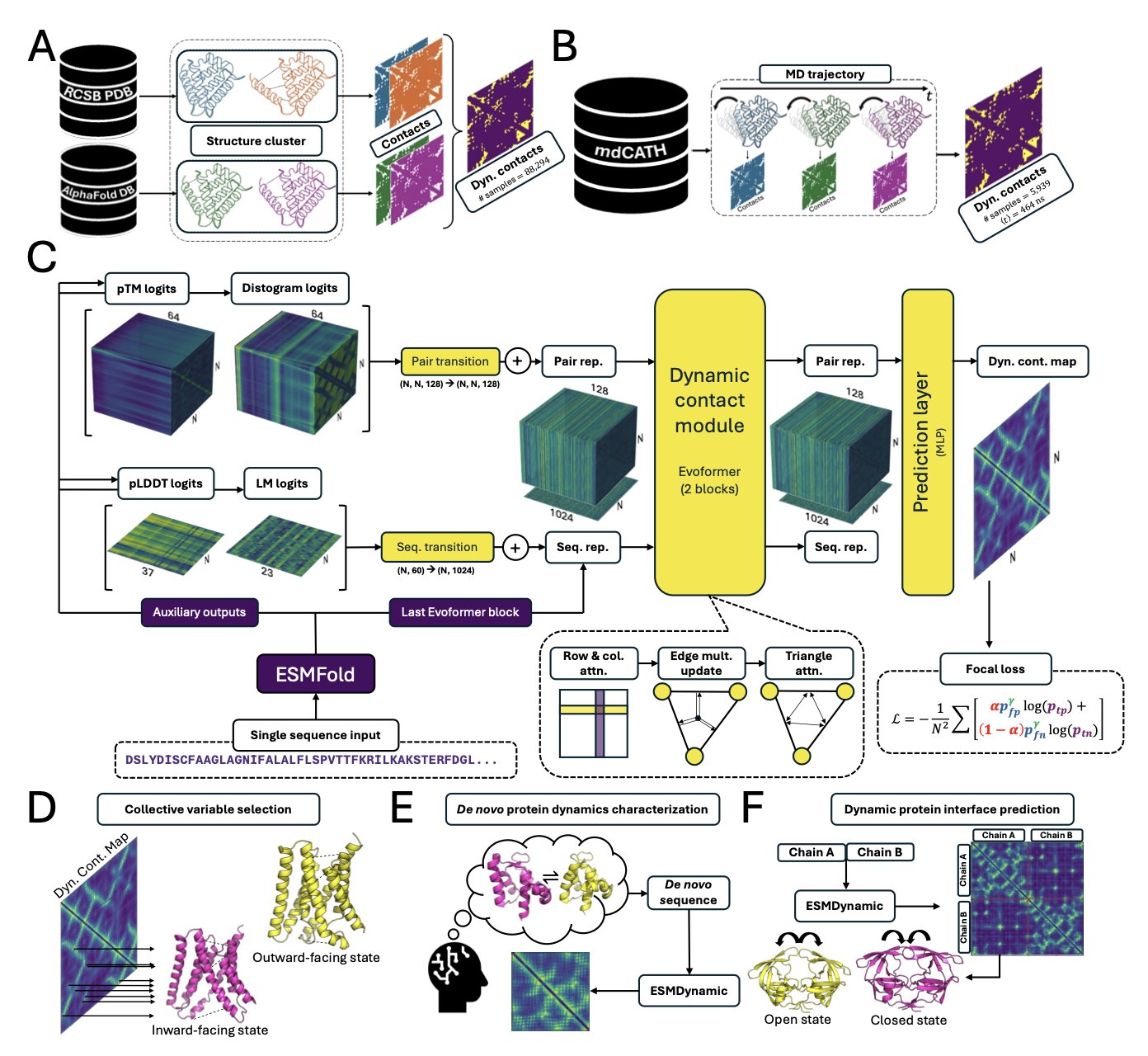
ESMDynamic 通过学习海量已有的蛋白质动态数据(一部分来自实验,一部分来自大规模 MD 模拟),掌握了蛋白质序列与动态行为间的规律,如同导演「阅片无数」后掌握了叙事技巧。它能识别出哪些氨基酸序列会构成柔性环区,或导致蛋白口袋的开合。
输入一条全新的氨基酸序列(「剧本」),ESMDynamic 就能利用学到的规律,快速预测出哪些氨基酸(「演员」)之间会发生频繁的动态接触。
实践检验
在两个大规模 MD 数据集上,ESMDynamic 的表现与需要更多输入的复杂模型(如 AlphaFlow)相当,但预测速度更快。
它在几个关键挑战上也表现出色:
ESMDynamic 提供了一个高效的「勘景工具」。在投入大量计算资源进行模拟前,可以用它快速定位蛋白质动态中值得关注的关键区域,预判「精彩剧情」的发生位置。
📜Title: ESMDynamic: Fast and Accurate Prediction of Protein Dynamic Contact Maps from Single Sequences
📜Paper: https://www.biorxiv.org/content/10.1101/2025.08.20.671365v1