目录
- 通过将文本、序列等多模态信息与图对比学习技术巧妙结合,这项研究为构建更强大、更具泛化能力的生物医学知识图谱提供了一套极具潜力的解决方案。
- MTAN-ADMET 通过强迫一个 AI 模型同时学习 24 种不同的 ADMET 性质,巧妙地解决了药物发现中数据稀疏的顽疾。
- 把分子结构图喂给视觉语言模型,再配上 SMILES 文本,确实能提升分子属性预测的准头,但真正的魔法在于如何聪明地进行微调。
- 迄今最大规模的分子语言模型研究 NovoMolGen 发现,模型的预训练指标和下游任务性能关联不大,且一个仅 3200 万参数的小模型,效果竟不输大模型。
- 这套名为 MMFRL 的新框架,通过一种「预训练作弊」手法和更精细的关系学习,让多种分子数据协同作战,把分子属性预测的准头和稳定性都提上了一个新台阶。
1. 知识图谱升级:多模态融合才是未来
知识图谱(KG)并不新鲜,它试图把药物、疾病、基因这些乱七八糟的点连成一张网,听起来很美。但现实是,大多数知识图谱都太「扁平」了。
一个叫「伊马替尼」的节点,在图里可能就只是个名字。可对做药的人来说,它是一串 SMILES 化学式,是一堆描述它激酶抑制谱的文献,还有无数的临床数据。只用一个名字,信息损失太大了。
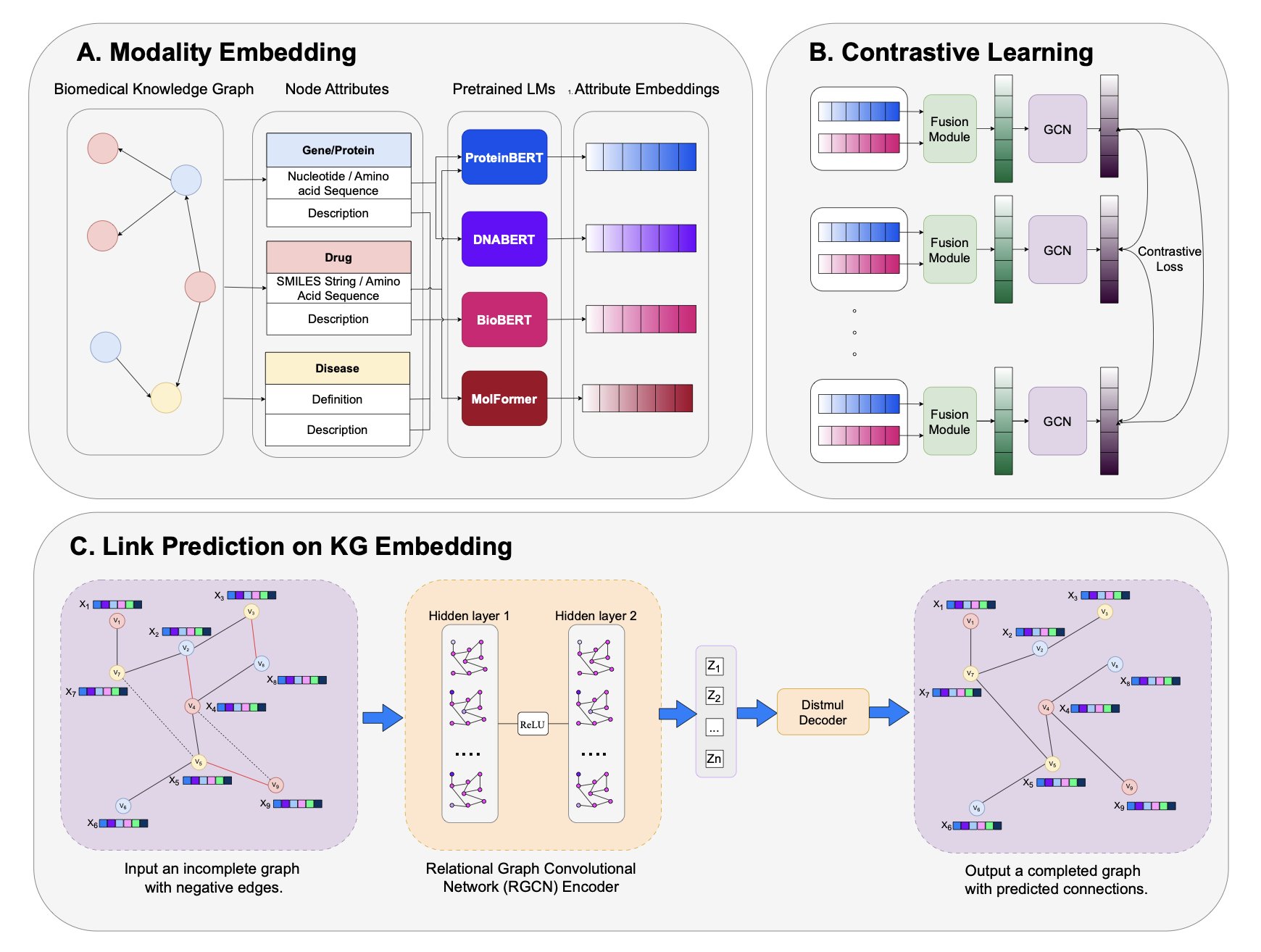
这篇新文章给知识图谱里的每个节点「充值」,注入多模态(Multimodal)信息。
他们干了啥?
首先,他们搞出来一个叫 PrimeKG++ 的增强版知识图谱。在这个图谱里,一个药物节点不仅有名字,还挂上了它的 SMILES 序列和详细的文字描述。同理,一个蛋白靶点,也连着它的氨基酸序列和功能注释。这就好比以前我们看的是黑白简笔画,现在直接升级到了 4K 高清彩色版。
有了这么好的数据,怎么用起来?
他们的流程分了几步走。第一步,用各种预训练好的语言模型(LMs)去「阅读」这些文本和序列信息,把它们都翻译成机器能懂的数学向量(嵌入)。
第二步,也是最关键的一步:「融合」。用一个注意力融合模块(比如 ReDAF),把来自不同模态的向量捏合成一个统一的、信息更丰富的节点表示。
光有丰富的节点还不够,节点之间的关系才是图谱的精髓。于是他们用了图对比学习(GCL)这把「刻刀」来精雕细琢。这技术听着玄乎,其实道理很简单。它告诉模型:「看,这个药和这个靶点在现实世界里是相互作用的,所以它俩的向量在数学空间里应该靠得近一点。那个药和这个靶点没关系,那就把它俩推远点。」通过成千上万次这样的「推拉」,整个图谱的结构就变得异常清晰、合理。
结果在预测药物 - 靶点、药物 - 疾病这些我们最关心的任务上,这套方法的表现非常扎实,尤其是在一些硬核的测试场景下,比如故意混入大量无关的「负样本」,它依然能火眼金睛地找出真正的关联。更重要的是,它对没见过的新药、新靶点同样有效。这才是 AI 模型能从实验室走向工业界应用的关键一步。
作者在文末也坦诚,他们的目的不是为了在某个排行榜上刷高零点几个百分点。我完全同意这种工作的价值在于为我们提供了一套更鲁棒、更可解释的工具。
当模型预测一个新的药物靶点时,我们或许可以追溯回去:这个判断主要是基于化学结构的相似性,还是某篇文献里的隐藏线索?
📜Title: Multimodal Contrastive Representation Learning in Augmented Biomedical Knowledge Graphs
📜Paper: https://arxiv.org/abs/2501.01644v2
💻Code: https://github.com/HySonLab/BioMedKG
2. MTAN-ADMET: AI 学会了「一心多用」
ADMET(吸收、分布、代谢、排泄、毒性)预测,一直以来都像是一场永无止境的、与「数据饥荒」的搏斗。
我们手里有几十个不同的 ADMET 终点要预测,但对于其中的绝大多数,我们拥有的、高质量的实验数据,少得可怜。有的性质,可能只有几百个数据点,而且其中「有毒」或「有问题」的分子,更是凤毛麟角。
在这种情况下,你试图去训练一个专门预测 hERG 毒性的 AI 模型,就像是想只通过看三张照片,就教会一个孩子认识什么是「猫」。他很可能会学到一些奇怪的、错误的规律,比如「猫就是白色的、毛茸茸的东西」。
MTAN-ADMET,就提出了一个解决方案:如果你想让一个孩子真正认识「猫」,你最好同时教他认识「狗」、「鸟」和「鱼」。
这个方法,就是「多任务学习」(Multi-Task Learning)。
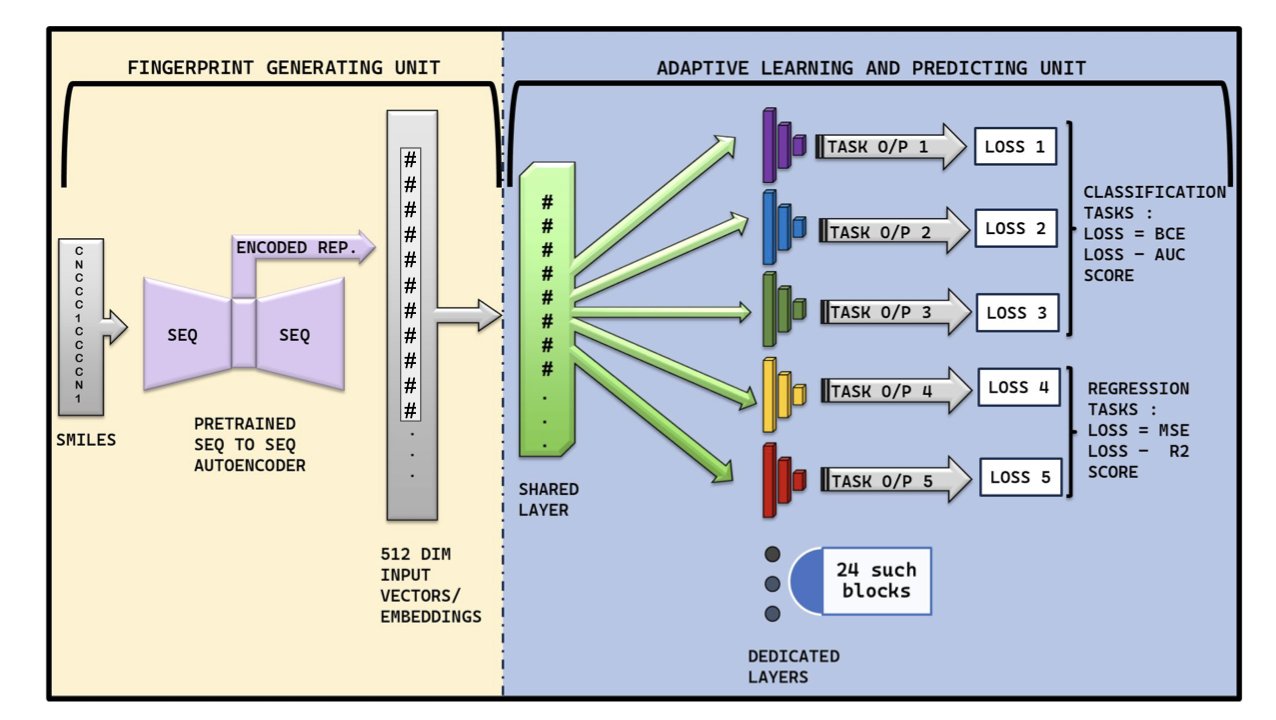
作者们没有去为 24 个不同的 ADMET 性质,分别训练 24 个独立的「专家」模型。相反,他们只训练了一个模型,但他们强迫这个模型,去同时学习所有这 24 件事。
这背后的逻辑是,虽然 hERG 毒性和 P450 抑制,在生物学上是两码事,但它们背后,可能共享了一些底层的、关于「一个分子,如何才能在一个复杂的生物系统里,举止得体」的化学物理规律。通过强迫模型去同时理解这两件事,它就被迫去学习那些更普适、更底层的化学知识,而不是去死记硬背某个特定数据集里的统计学巧合。
当然,想让一个模型同时学会 24 件不同的事,而且这些事里,有的需要它给出一个「是/否」的答案(分类,比如有没有毒),有的又需要它给出一个具体的数值(回归,比如溶解度是多少),这在技术上,无异于让一个人同时参加 24 场不同规则的奥运会项目。
这正是 MTAN-ADMET 的精妙之处。它的架构里,包含了一些非常「自适应」机制:
这个「通才」模型的表现如何?
结果是,它在 24 个 ADMET 性质中的 14 个上,都击败了现有的「专家」模型。尤其是在预测心脏毒性这类出了名难搞、数据又少又偏的问题上,它表现得非常好。更重要的是,它做到这一切,仅仅是靠「阅读」最简单的 SMILES 分子字符串,而无需那些计算上更昂贵、处理起来也更麻烦的图结构。
所以,MTAN-ADMET 的价值,不仅仅在于它又是一个性能不错的预测工具。它的价值在于,它为每天都在和稀疏、混乱、不平衡的生物学数据作斗争的人,指明了一条非常重要的、可能也更接近生物学现实的道路:有时候,想成为一个更好的专家,你需要的,可能恰恰是去学一些看似无关的东西。
📜Title: MTAN-ADMET: A Multi-Task Adaptive Network for Efficient and Accurate Prediction of ADMET Properties
📜Paper: https://doi.org/10.26434/chemrxiv-2025-zhrsk
Code: https://github.com/TeamSuman/MTAN-ADMET
3. MolVision:AI 看图识分子,化学预测新范式
用 SMILES 字符串来教 AI 认识分子,有点像在自欺欺人。
任何一个有机化学家都知道,一个简单的文本串根本无法捕捉到一个分子的全部精髓——那些微妙的立体化学、环张力,或者某个官能团在空间中的朝向。这些东西,我们看一眼 2D 结构图就心领神会了。这才是化学家思考的方式。
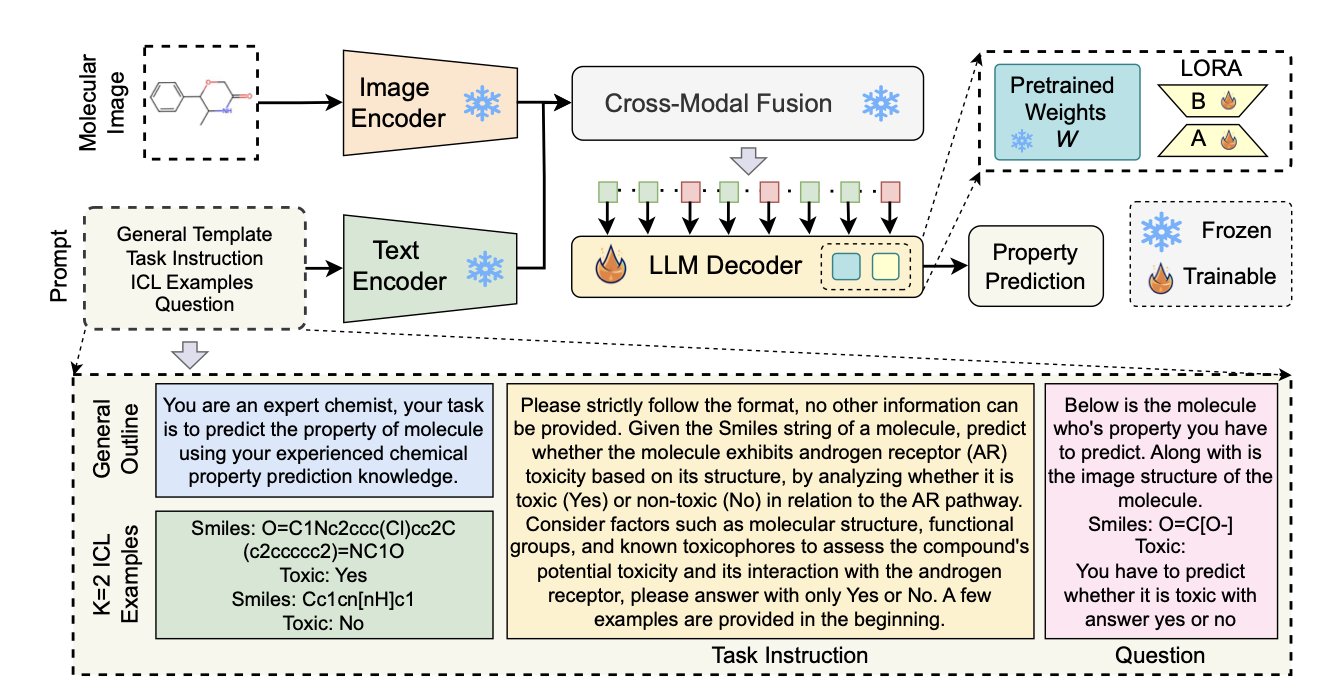
MolVision 感觉就像是 AI 终于开始学着讲「人话」了,或者说是「化学家的话」。研究者的思路是:既然图片信息这么重要,为什么不直接让模型看图呢?他们把分子结构图和 SMILES 文本一起打包,丢给了最先进的视觉语言模型(VLM)。
但事情没那么简单。光有图还不行,直接把预训练好的 VLM 拿来用,效果很可能一团糟,因为这些模型是看猫、看狗、看风景训练出来的,它们压根不「懂」化学键和原子是什么。
这里的关键一步是微调。
作者们发现,LoRA 这种低秩适配技术简直是天赐之物。它能在不触动整个庞大模型的前提下,高效地教会模型如何解读化学结构。这就好比给一个语言天才一本化学图解词典,让他快速入门。再加上一种对比学习策略——本质上是让模型反复比较,「这两个分子看起来像,因为它们都有一个苯环」或者「这两个分子不一样,一个有手性中心」——视觉编码器才算真正开了窍。
结果不出所料,但又在情理之中。
图像加文本的组合,在毒性、溶解度、生物活性等一系列属性的预测上,表现确实比单用文本要好。更重要的是,模型的泛化能力变强了。这意味着它在面对一个全新骨架的分子时,不会那么容易「懵圈」,而是能从学到的视觉模式中做出更靠谱的推断。
这当然不是说我们马上就能扔掉所有的物理化学计算了。但这项工作提供了一个坚实的基准,证明了把化学家最直观的工具——2D 结构图——融入 AI 模型是条完全走得通的路。
📜Title: MolVision: Molecular Property Prediction with Vision Language Models
📜Paper: https://arxiv.org/abs/2507.03283v1
💻Code: https://molvision.github.io/MolVision/
4. NovoMolGen:分子大模型,小也有小的好
我们似乎已经习惯了一个「信条」:模型越大,数据越多,效果就越好。从 GPT-3 到 GPT-4,参数量一路飙升,大家都在追求「大力出奇迹」。
这股风气也刮到了化学信息学领域。分子语言模型(Mol-LLMs)也越做越大。
但在分子这个特殊的、有明确物理化学规则的世界里,真的是「越大越好」吗?
这篇论文的 NovoMolGen 研究,就对这个问题,做了一次迄今为止最大规模、也最系统性的「拷问」。
他们在一个包含 15 亿个分子的超大数据集上,训练了一系列不同大小的 Transformer 模型,试图摸清分子语言模型的「脾气」。
结果得出了几个非常「反常识」,但极具指导意义的结论。
发现一:预训练的「期末考分」,参考价值不大
我们训练一个语言模型,通常会看一些指标,比如困惑度(Perplexity),来评估它学得怎么样。就像一个学生的期末考试分数。
但 NovoMolGen 的研究发现,一个模型在预训练阶段的「考分」,和它毕业后在实际工作(下游的分子生成任务)中的表现,相关性惊人地弱。
这意味着,我们过去可能花了大量的计算资源,去把一个模型的「考分」从 95 分刷到 96 分,但这对它解决实际问题的能力,可能并没有什么实质性的帮助。这和通用的自然语言处理(NLP)领域很不一样,也提醒我们,不能把 NLP 的经验,想当然地照搬到分子世界里来。
发现二:性能很快就「饱和」了
更令人惊讶的是模型的性能,在训练过程的早期阶段,就已经基本达到了「天花板」。再往后继续砸钱、砸时间去训练,带来的性能提升已经微乎其微。
好比一个运动员,通过前三个月的魔鬼训练,百米成绩已经从 12 秒提高到了 10 秒 5。再往后训练一年,可能也只能提高 0.1 秒。对于追求「性价比」的药物研发项目来说,我们需要认真思考,这额外的投入,到底值不值。
发现三:「小个子」也能是「优等生」
这项工作最有价值的成果,就是他们推出的 NovoMolGen 模型家族,特别是其中那个只有 3200 万参数的「小个子」模型。
在多个分子生成的基准测试中,这个小模型的表现,与那些比它大几倍甚至几十倍的模型相比,毫不逊色,甚至在某些任务上还更胜一筹。
这意味着,我们不需要动辄就上 A100 集群,可能在一台性能不错的单机上,就能部署一个足够强大的、能解决实际问题的分子生成模型。这大大降低了 AI 辅助药物发现的技术和成本门槛。
NovoMolGen 这项研究,让我们对分子语言模型的真实能力和训练规律,有了更清醒的认识,在分子设计这个领域,与其盲目地追求「大」,不如更聪明地去追求「好」和「巧」。
📜Paper: https://arxiv.org/abs/2508.13408v1
💻Code: http://github.com/chandar-lab/NovoMolGen
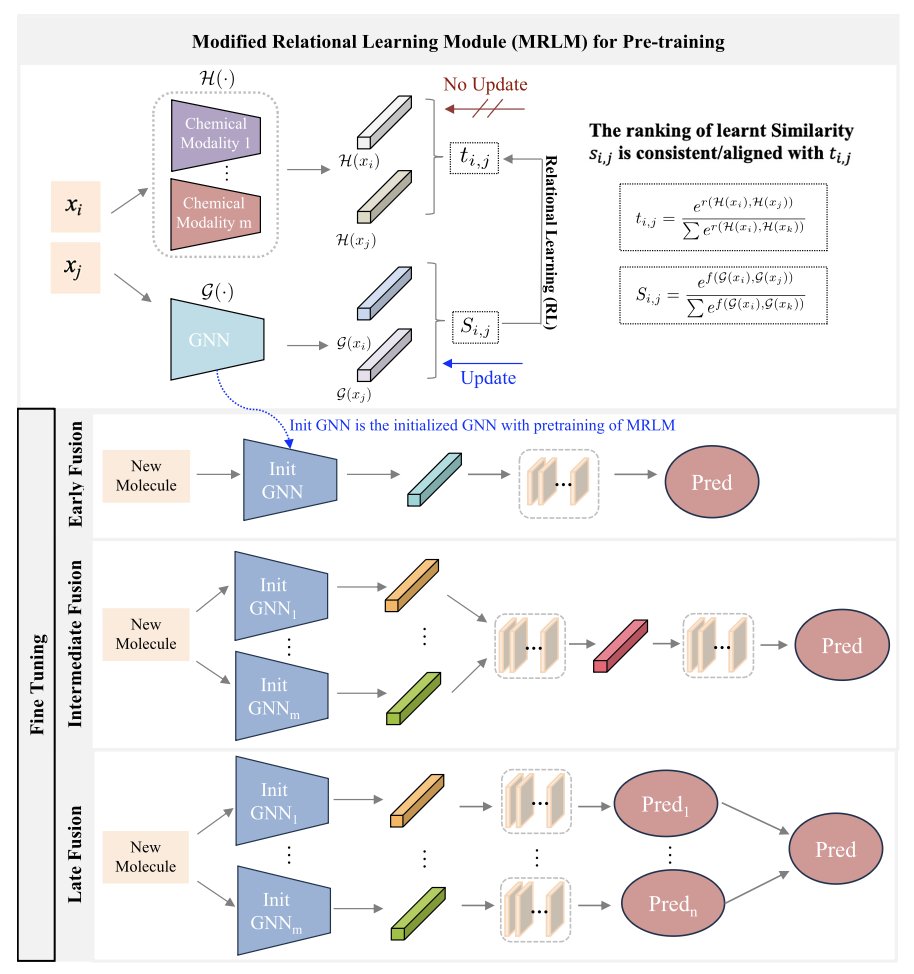
5. MMFRL 模型:AI 预测分子,这次能玩出什么新花样?
我们手里的分子数据,从来都不是单一维度的。你有 2D 结构图,有 SMILES 序列,有 3D 构象,可能还有一大堆零散的实验数据。就像做菜手上有牛肉、土豆、胡萝卜和红酒。问题是,你怎么把它们做成一道法式红酒炖牛肉,而不是一锅东北乱炖?
这就是「多模态融合」要解决的难题。简单地把所有数据一股脑扔进模型?通常效果很糟。
MMFRL 框架处理这个问题的手法很务实。研究者系统地比较了「早期融合」(一开始就把所有食材扔锅里)、「中期融合」(各自处理一下再混合烹饪)和「晚期融合」(各做各的菜,最后再拼盘)的策略。结果发现,「中期融合」在大多数任务上表现最好。这不意外,但系统性地证明这一点,本身就很有价值。
「预训练时利用辅助模态」的操作是什么意思?打个比方,这就像一个医学生,在学校里不仅学了核心课程,还读了大量病例、看了很多手术录像(这些就是辅助模态)。等他毕业当了医生(进入推理阶段),就算手头没有那些教科书和录像了,他从中学到的经验和直觉已经内化,让他能做出更准确的诊断。
MMFRL 就是这么干的:它在「上学」阶段,用尽可能多的数据类型来训练自己,哪怕这些数据在「毕业上岗」时根本用不到。这是一种极其办法,能把宝贵的信息榨干,塞进模型的「大脑」里,又不增加最终预测的计算负担。
另一个核心是所谓的「关系学习」。传统的模型看分子,要么是孤立地看,要么是两两对比看「像不像」。MMFRL 更进一步,它学的是「关系的关系」。它不只关心 A 和 B 的关系,还关心「A 与 B 的关系」和「C 与 D 的关系」这两组关系之间是否相似。这就像理解一个人,不光看他的个人档案,还要看他的社交网络,看他和他朋友的互动模式。这种更高维度的视角,显然能捕捉到更复杂、更微妙的化学规律。
当然,按照惯例论文总要说它在 MoleculeNet 这个标准竞技场上「拳打南山敬老院,脚踢北海幼儿园」,击败了所有 SOTA (state-of-the-art) 模型。这很好,说明这套组合拳确实有效。
但在工业界更关心的是:它是不是又一个黑箱?好在作者们也考虑到了这一点,展示了模型的可解释性分析。能让我们知道模型在做预测时,究竟盯上了分子的哪个部分,这对于设计新分子至关重要。一个无法解释的预测,和一个随机数生成器有多大区别?
所以,MMFRL 是一次漂亮的工程实践。它直面了分子数据 messy(杂乱)且常常不完整的现实,尤其是那个「借用未来数据训练」的点子非常有效。接下来,就看它能否走出基准测试的舒适区,在真实的药物发现项目中帮我们找到点什么了。
📜Title: Multimodal fusion with relational learning for molecular property prediction
📜Paper: https://www.nature.com/articles/s42004-025-01586-z
💻Code: https://github.com/zhengyjo/MMFRL