目录
- 用 nanoGPT 作为 AI 智能体优化代码的基准,这个想法很棒,但结果也给“AI 自我进化”的狂热泼了盆冷水:AI 目前还差得远。
- 一个系统同时搞定文献的“影响力”和“相关性”,再用两个 AI 给你写摘要,文献综述的苦日子可能到头了。
- MDZip 用神经网络实现了超过 95% 的分子动力学数据压缩,代价是物理保真度上的些许妥协,但这对于数据归档和共享而言,绝对是笔划算的买卖。
1. nanoGPT 基准:AI 写代码,离谱还是靠谱?
如果你进场刷推,你可能已经听说或“体验”过 Andrej Karpathy 的 nanoGPT。该项目最初只是一个极简的 GPT 训练教程。然而,优质的项目往往能够自我发展,nanoGPT 自然也不例外。
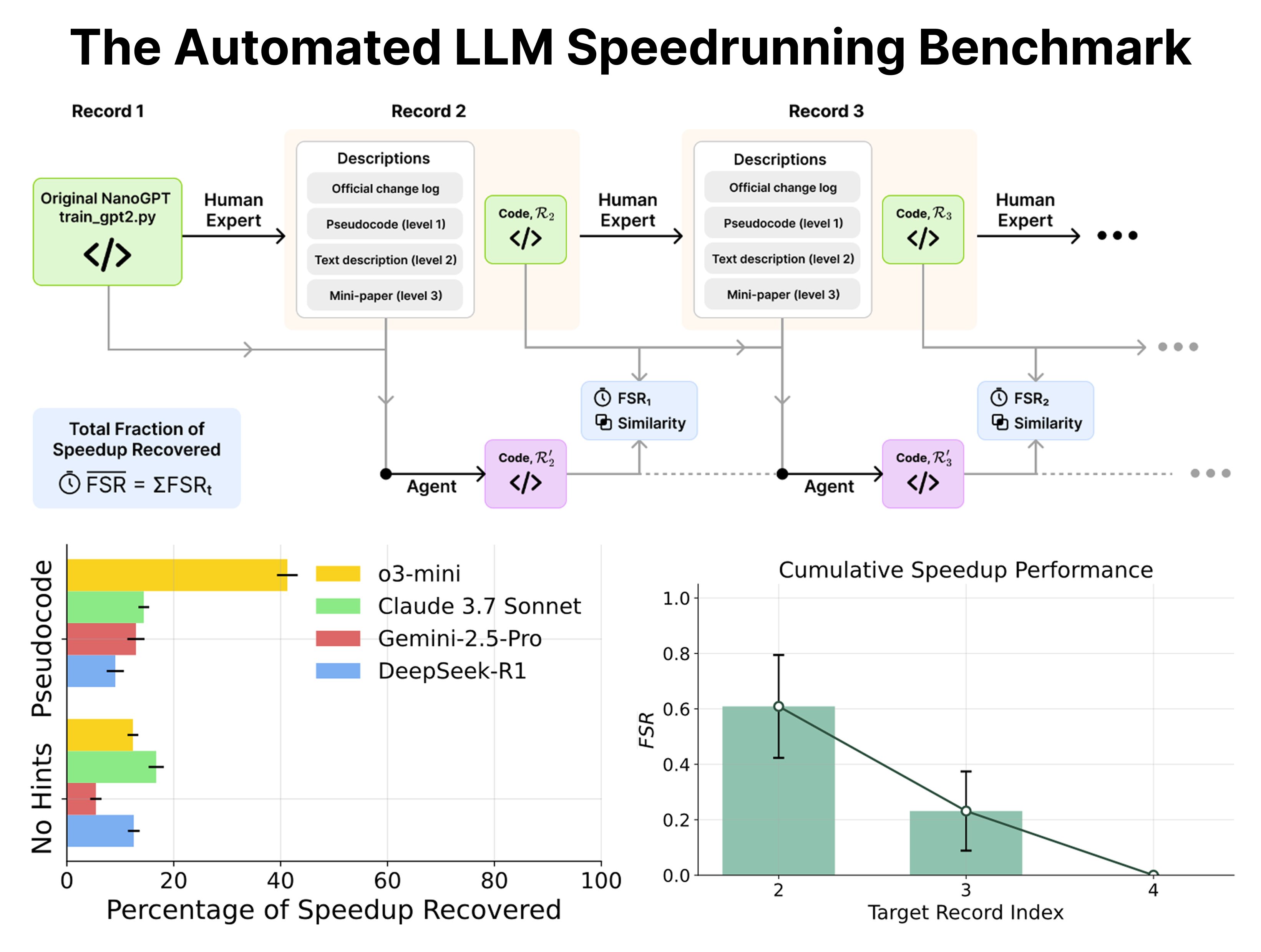
一开始,Karpathy 通过它向大家展示了如何从零开始训练 GPT。随后,他又使用 C 语言和 CUDA 对其进行了复刻,nanoGPT 因而成为新项目的基准。之后,社区中的专家们开始将其作为“研究沙盒”进行创新改进,把 GPT-2 (124M) 模型的训练时间,从最初的 45 分钟压缩至现今的 3 分钟!这一成就非常具体且可量化,是人类智慧的成果。
现在,最精彩的部分来了。
有了这么一个清晰的“人类记录”,nanoGPT 顺理成章地变成了一个完美的基准测试:既然人类能把代码优化到这个程度,那让现在最火的 LLM 智能体来试试怎么样?给它不同程度的提示,看它能把训练速度提高多少?
这篇新论文就干了这个事。剧透一下:结果不太乐观。
哪怕有明确的提示,AI 智能体的表现也只能说是差强人意。这就像你告诉一个机器人“把这辆车的百公里加速从 10 秒提升到 5 秒”,它顶多给你换个轮胎,而人类工程师已经把发动机、变速箱和空气动力学都重新设计了一遍。
这自然就引出了那个经久不衰的话题:递归式自我改进(Recursive Self-improvement)。很多人一听到这个词,脑子里就浮现出“天网”或者“Llama 5 一夜之间造出 Llama 6”的科幻场景。
Karpathy 的观点一向很实在:这种自我改进根本不是什么“开关一开,奇点降临”的玩意儿。它早就开始了,而且是以一种我们习以为常、平滑渐进的方式。
想想看,你用的 IDE、代码补全工具、Google 搜索,甚至 Git,不都是在加速你构建下一个版本的软件吗?这些工具,就是“智能”在辅助我们迭代。现在,我们工具箱里多了个更强大的扳手——LLM,我们开始跟它协作完成更大块的功能,这种协作只会越来越深入。这才是现实。
而且,我们必须对问题的复杂度有清醒的认知。nanoGPT 整个项目也就 750 行代码,还只涉及预训练阶段。这在真实世界里是什么概念?这就像是在一个标准卡丁车赛道上测试车辆性能。而我们工业界的生产级代码库,动辄几十上百万行,其复杂程度好比要跑完勒芒 24 小时耐力赛,还得自己处理各种突发状况。两者之间差了好几个数量级。
把 nanoGPT 作为当前 AI 能力的一个标尺,是再合适不过了。它足够简单,让我们能在一个可控的环境里,戳一戳 AI 的真实水平;它又足够有挑战性,能暴露出 AI 在逻辑推理、规划和代码优化这些核心能力上的短板。
📜Title: Can Large Language Models Automate Research? A Case Study on nanoGPT
📜Paper: https://arxiv.org/abs/2405.17387
2. 双 LLM 模型:你的下一个科研文献神器?
我们每个人都清楚,在信息的汪洋里找几篇真正有用的文献是什么感觉——就像拿着一个漏勺想从消防水管里喝水。每周都有成千上万篇新论文冒出来,大部分你永远都不会读,剩下的那些,光是筛选和通读摘要就足以耗尽你的咖啡因储备。
所以,当看到有人想用 AI 来正经解决这个问题时,我总是既期待又怀疑。这篇预印本里的系统,坦白说,有些想法还真挺对一线研发人员的胃口。
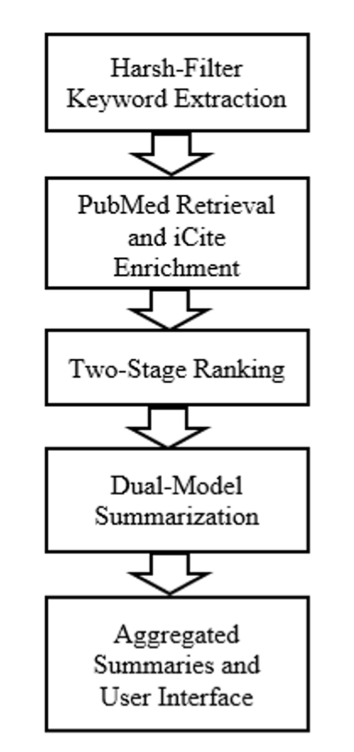
首先,他们搞的这个排序算法有点意思。我们都用过 PubMed 或者 Google Scholar,要么是按引用数给你一堆“上古神文”,要么是按关键词匹配给你一堆不知所云的新东西。这个系统试图走中间路线,它把一个叫“相对引用率”(RCR)的指标和传统的“余弦相似度”结合起来。简单说,它不只关心这篇论文有多少人引用(学术影响力),还关心它的内容是不是真的跟你搜的东西八竿子打得着(主题相关性)。这个组合拳打得不错,理论上能帮你过滤掉那些“名气很大但与你无关”和“看似相关但毫无分量”的论文,让你拿到的列表质量更高。
更有趣的是摘要部分。他们没押宝在任何一个大模型上,而是同时用了 Google 的 Gemini 2.0 和 OpenAI 的 GPT-4o-mini。这招很聪明。我们都知道,现在没有哪个 LLM 是完美的。有的擅长抓大放小,有的则在细节上抠得更准,但它们也都可能一本正经地胡说八道(也就是“幻觉”)。让两个模型同时上阵,就像是给你的实验数据找了两个人独立分析。结果相互印证,能极大地提高摘要的准确性和可靠性,还能减少单一模型犯傻的概率。对于需要从摘要里快速抓住方法学和关键结论的人来说,这简直是救命稻草。
当然,光说不练假把式。他们跑了 20 个生物医学领域的查询,BERT-F1 分数平均有 0.86。这个分数不算惊为天人,但绝对是“优秀”级别了,说明机器判断的相关性跟人差不太多。不过,我更看重的是那 10 个用户的真人反馈。平均分超过 4.5/5,这可不是刷出来的。用户特别提到,这个系统生成的摘要能清晰地标出“研究方法”和“作者单位”,这可是我们行内人最关心的信息之一。它知道我们想看什么,这比单纯的文字压缩重要得多。
作者们还提到,想把这套方法推广到其他领域,并且加入“自适应智能”和“隐私保护”。这听起来很宏大。不过,从生物医学跨到,比如说,高能物理或者有机化学,知识图谱和术语体系完全是两码事,挑战不小。至于隐私,当你把研究思路作为查询喂给商业公司的 API 时,谈隐私总是有点……乐观。
📜Title: Multi-Model LLM Architectures for Personalized Summarization and Relevance Ranking in Biomedical Literature
📜Paper: https://www.biorxiv.org/content/10.1101/2025.07.29.667503v1
3. MDZip:给你的分子动力学模拟数据来一场终极瘦身
做分子动力学(MD)模拟的,谁没有被数据存储搞得焦头烂额?一次模拟生成的轨迹文件轻易就占用几个 TB 的硬盘空间,让你在“删还是不删”的危机中苦苦挣扎。现在,有人想利用神经网络为你的数据进行一次“抽脂手术”。
这篇论文中的 MDZip 正是这个概念的体现。
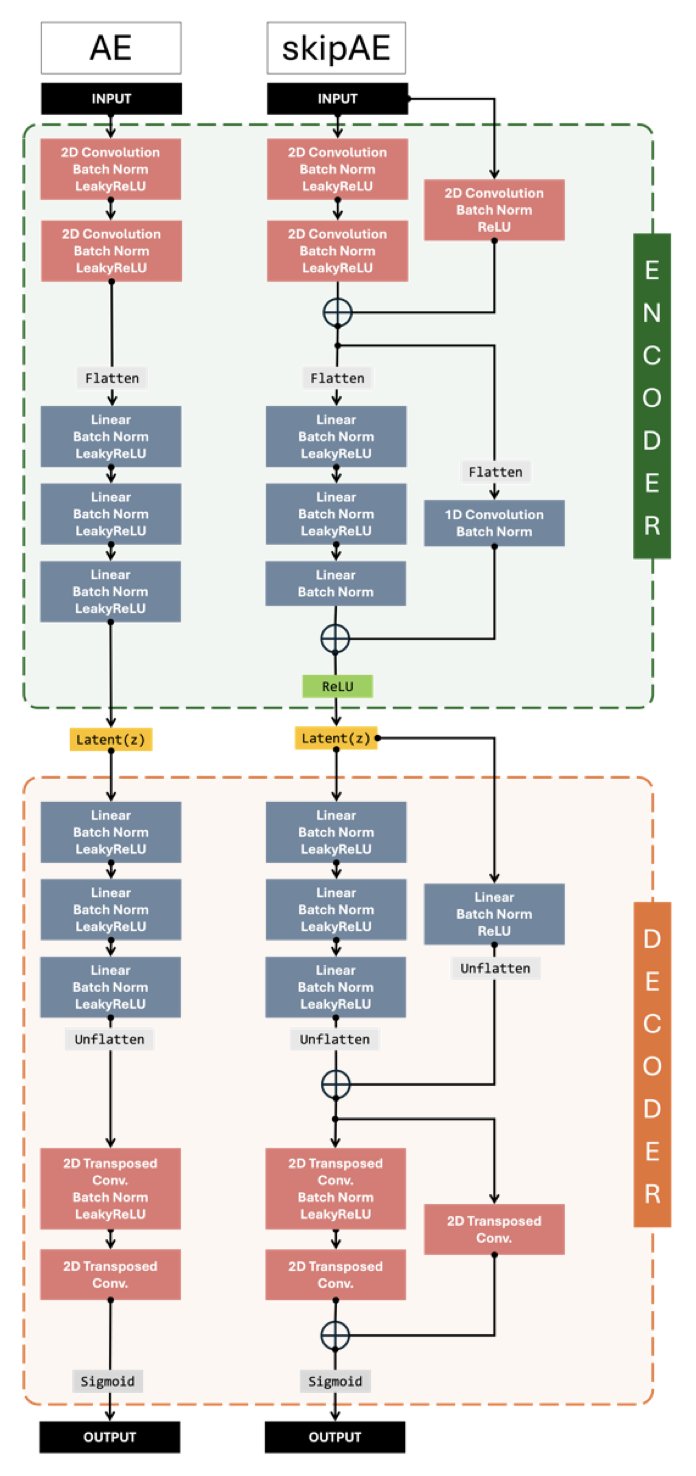
简单来说,它是一个基于卷积自编码器(Convolutional Autoencoder)的压缩框架。你可以将其视为一个极其聪明的“速记员”。你不仅不需要记录蛋白质每时每刻的所有原子坐标,而是通过训练这个速记员,使其掌握一套针对特定体系的独特简写方法(即“紧凑的潜在表示”)。当需要回顾轨迹时,它可以将简写“翻译”为完整的原子坐标。
结果怎么样?
相当惊人。压缩率超过 95%!这意味着以前需要 20 块硬盘才能存下的数据,现在 1 块就够了。对于大规模模拟和数据共享来说,这简直是久旱逢甘霖。
“听起来太美好了,代价是什么?”
这个方法的精髓在于它是一个“残差自编码器”(Residual Autoencoder)。它儿比传统的自编码器要聪明一点。传统的自编码器是看着原始图像(或结构)从零开始画一幅复制品;而残差自编码器更像是先打个草稿,然后专门去学习和修正草稿与原图之间的“差值”。这种“专注于修正错误”的模式,让它在重建结构时精度更高,离谱的构象也更少。从结果来看,无论是 RMSD 涨落,还是距离分布这些我们常看的系综性质,MDZip 都保留得不错。
但这里有个关键点,MDZip 是“物理不可知”(physics-agnostic)的。它的神经网络并不懂什么是范德华力,什么是键长键角。它只关心一件事:用最紧凑的方式编码坐标,并且能尽可能精确地解码回来。这既是优点也是缺点。优点是它通用性强,蛋白质、核酸、复合物都能用。缺点就是,它重建出来的构象可能“看起来很美”,但在能量上却不那么合理。比如,两个原子可能靠得太近,产生了不该有的碰撞。
这就引出了最大的代价:能量保真度的损失。如果你想用解压后的轨迹去做结合自由能计算(比如 MM/PBSA),那你可得小心了。一个在能量上不合理的构象,可能会让你的计算结果错得离谱。
研究者也意识到了这个问题,并给出了一个补救措施:对解压后的构象做一次短暂的能量最小化。这就像把一件在行李箱里压皱了的衣服拿出来用蒸汽熨斗过一遍。它能抚平那些最扎眼的“褶皱”(比如原子碰撞),让结构恢复到物理上比较合理的状态。但这终究是个“补丁”,它能让构象变得“合理”,但无法保证它就是模拟中那个“原始”的构象。
所以,MDZip 到底是不是个好东西?
看你怎么用。如果你只是想归档海量的模拟数据,或者把轨迹分享给合作者做一些宏观的结构分析(比如看蛋白是怎么运动的,或者某个 loop 区是不是很柔性),那它是神器。它用一点点能量保真度的牺牲,换来了存储便利。但如果你要做的分析对能量细节极其敏感,那最好还是老老实实地用原始轨迹。
📜Title: MDZip: Neural Compression of Molecular Dynamics Trajectories for Scalable Storage and Ensemble Reconstruction
📜Paper: https://www.biorxiv.org/content/10.1101/2025.07.31.667955v1